
Fizyczka, chemiczka, kierowniczka Interdyscyplinarnego Laboratorium Modelowania Układów Biologicznych w Centrum Nowych Technologii Uniwersytetu Warszawskiego. Zainteresowania badawcze: modele teoretyczne krajobrazu energetycznego białek, metody analityczne i narzędzia bioinformatyczne w przewidywaniu struktur białkowych, właściwości mechaniczne białek, degradacja i translacja.
Białka to podstawa. Są w każdej komórce naszego ciała i pełnią rozmaite funkcje. Budują, transportują, odbierają sygnały, powodują ruch… W uproszczeniu to takie sznurki składające się z aminokwasów. Ale – co fascynujące – sama informacja o tym, z jakich aminokwasów składa się białko, nie wystarcza do zrozumienia, jaką funkcję pełni. Potrzebna jest wiedza o jego przestrzennej strukturze.
I teraz: skąd to „wow” w tytule?
To jest tak ważne, że istnieje specjalny konkurs CASP (Critical Assessment of Protein Structure Prediction) na przewidywanie struktury 3D białek. Najpierw zespoły doświadczalne starają się otrzymać ich strukturę w laboratorium. To, co im wyjdzie, jest wzorem. Następnie grupy teoretyczne próbują na podstawie sekwencji (kolejności) aminokwasów przewidzieć, jak białko wygląda w trzech wymiarach. Następnie porównuje się wyniki.
Pod koniec zeszłego roku AlphaFold 2, program firmy DeepMind (kupionej przez Google’a), zostawił konkurencję w CASP daleko w tyle. Program potrafił przewidzieć ze znakomitą dokładnością (ok. 90% zgodności) strukturę 2/3 białek. – To było to wow! – mówi w podcaście dr hab. Joanna Sułkowska, prof. Uniwersytetu Warszawskiego, kierująca Interdyscyplinarnym Laboratorium Modelowania Układów Biologicznych.
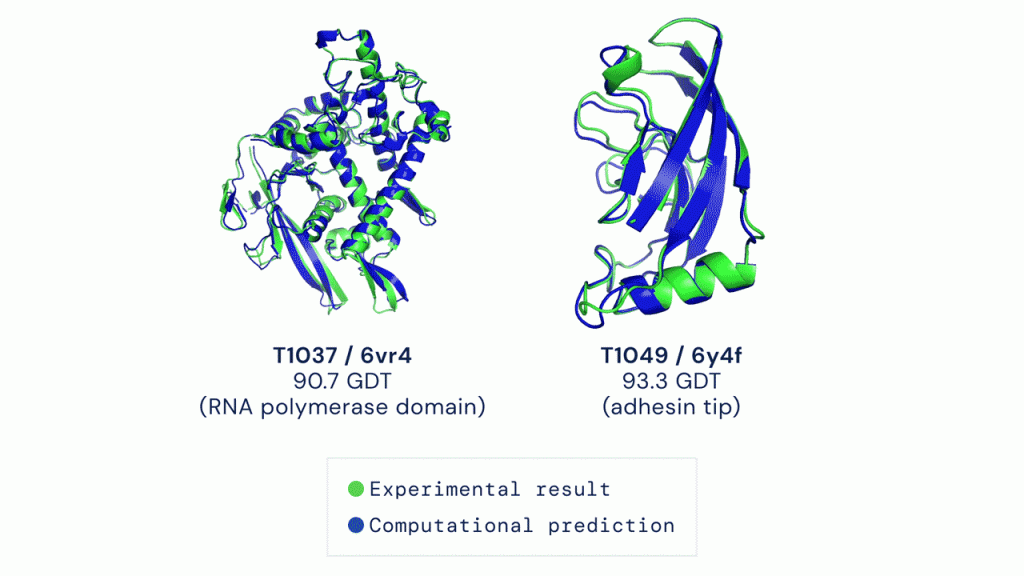
Zobaczcie przykład, kolor zielony to wynik z laboratorium, niebieski z obliczeń programu:
Dlaczego to ważne? Jeśli będziemy dysponować oprogramowaniem, które będzie szybko i dobrze przewidywało strukturę białek, nie będziemy musieli żmudnie jej sprawdzać w laboratoriach (albo będziemy mogli robić to rzadziej). Będzie sprawniej i taniej, co może się przełożyć na lepsze opracowywanie leków. Albo pozwoli na stworzenie enzymu zjadającego plastik. I wiele innych możliwości. To może być naprawdę krok w stronę rewolucji.
W tym filmiku DeepMind zgrabnie to wyjaśnia:
Ten odcinek jest dla mnie szczególną przyjemnością, bo mogę w nim porozmawiać z jedną z tych badaczek, które zrobiły na mnie największe wrażenie. Jest mądra, konsekwentna, zdolna, pracowita i pracuje na pograniczu biologii, fizyki i chemii. Zaprawdę, do takich osób należy przyszłość nauki. Musicie koniecznie posłuchać prof. Sułkowskiej!
Obrazek ilustracyjny: białko hemoglobina, Wikipedia; Richard Wheeler (Zephyris) 2007. Created with en:pymol from en:PDB enzyme
TRANSKRYPCJA
Karolina Głowacka: Dzień dobry. Cieszę się, że jesteście. Więcej o projekcie i o mnie przeczytacie na radionaukowe.pl, a jeśli spodoba wam się ten podcast, możecie go wesprzeć na patronite.pl/radionaukowe. Wszystkim patronkom i patronom bardzo dziękuję.
„To zmieni wszystko” – głosił nagłówek w „Nature”. Pod koniec zeszłego roku sztuczna inteligencja w postaci programu AlphaFold 2 przewidziała strukturę 3D białek w niespotykanej do tej pory skuteczności. Naukowcy zyskali potężne narzędzie, które może pomóc m.in. w szybkim opracowywaniu leków. Czy mamy do czynienia z rewolucją w biologii? Radio Naukowe, odcinek trzynasty. Zaczynamy.
Białka są w każdej komórce naszego ciała i pełnią rozmaite funkcje – budują, transportują, odbierają sygnały, powodują ruch, są absolutnie fundamentalne. To takie sznurki składające się z aminokwasów. Ale – to fascynujące – sama informacja o tym, z jakich aminokwasów składa się białko, nie wystarczy, żeby rozumieć jego funkcję. Musimy jeszcze precyzyjnie wiedzieć, jak naturalnie się skręca, układa. Dlaczego tak jest? Za chwilę usłyszycie w podcaście. Do wyjaśnienia tej sprawy i całego zamieszania związanego z AlphaFold 2 zaprosiłam badaczkę, która udowodniła światu, że białka potrafią się wiązać w supełki. To jest bardzo poważna sprawa, może mieć znaczenie dla wyjaśnienia np. choroby Alzheimera. W internecie możecie znaleźć filmy, na których ta badaczka ze sznurkiem w dłoni tłumaczy, w czym rzecz. Nie mówiłam jej tego, ale moja dzisiejsza rozmówczyni autentycznie mnie onieśmiela, chociaż znamy się od kilku lat. Jest zdolna, mądra, pracowita, uparta i łączy wiedzę z fizyki, chemii czy biologii. Do takich należy przyszłość nauki. Posłuchajcie.
***
K.G.: Doktor habilitowana Joanna Sułkowska, profesor Uniwersytetu Warszawskiego, kierownik Interdyscyplinarnego Laboratorium Modelowania Układów Biologicznych. Dzień dobry, Joanno.
Joanna Sułkowska: Dzień dobry, Karolino. Dzień dobry, państwu.
K.G.: Powiedz, czego w zasadzie udało się dokonać AlphaFold, że wywołało to aż takie emocje? Bo rozumiem, że w twoim środowisku badaczy zajmujących się białkami to musiało wywołać emocje.
J.S.: To było jednym słowem przełomowe wydarzenie, a jednym zdaniem, dlaczego było przełomowe? Ponieważ rozdzielczość, którą otrzymały zespoły, konkretnie AlphaFold 2 jest porównywalna z rozdzielczością obecnych wyników eksperymentalnych, czyli udało się przewidzieć jakościowo strukturę białka z dokładnością, którą otrzymujemy obecnie z eksperymentu, więc jest to przełom. Możemy już odstawić pracochłonne badania doświadczalne, ponieważ można powiedzieć, że teoria przekroczyła granicę eksperymentu.
K.G.: Czyli zamiast spędzać tygodnie w laboratorium, narażając swoje kręgosłupy na wygięcia i problemy, można po prostu w pewnym sensie na sucho dzięki wykorzystaniu komputerów przewidzieć tę strukturę przestrzenną białek, tak? Różnica jest w tempie? I pewnie też w kosztach takiego przewidywania?
J.S.: Tak, ale tu jeszcze nawet można pokusić się o powiedzenie, że nawet w kilku przypadkach wyniki teoretyczne pozwoliły lepiej otrzymać strukturę doświadczalnie. Może jeszcze jedno zdanie wytłumaczenia, że AlphaFold brał udział w tzw. międzynarodowych zawodach do przewidywania struktury białek, które się teraz odbywały po raz czternasty. Odbywają się one co dwa lata i ich założeniem jest, że zespoły doświadczalne na całym świecie, które są zaangażowane w takie wspólne konsorcjum, starają się otrzymać strukturę doświadczalnie, czyli np. za pomocą metod krystalograficznych czy metod NMR. I te wyniki są niejawne. Następnie grupy teoretyczne mają jakiś czas, żeby spróbować na podstawie tzw. sekwencji aminokwasów przewidzieć, jak ta sekwencja wygląda w trzech wymiarach, czyli jak wygląda tzw. fold białka. Fold białka to taka struktura, która pełni funkcję biologiczną, która jest aktywna biologicznie.
K.G.: Fold, czyli takie skręcenie, złożenie?
J.S.: Tak, takie trójwymiarowe upakowanie przestrzenne. Czyli mogą powstać tzw. spinki albo helisy. Jest kilka takich elementów, które się razem przestrzennie trzymają za pomocą różnych oddziaływań elektrostatycznych. Myślę, że każdy z nas wie, czym są takie podstawowe oddziaływania elektrostatyczne. Zadaniem teoretyków było do tej pory spróbować otrzymać podobny fold. Bo taką sekwencję można wziąć do ręki i zgnieść, i to już jest taki trójwymiarowy fold. Ale całe wzywanie polega na tym, żeby to był ten, który ewolucja sobie wymyśliła, że dana sekwencja będzie miała ten jedyny, unikalny fold, który z kolei będzie aktywny biologicznie. Tu jeszcze jedno zdanie wytłumaczenia – na koniec trzeba ocenić, na ile ktoś coś dobrze zrobił. Taką miarą jest np. miara liczbowa, nazywa się to global distance. Zaczyna się od zera i kończy na stu. Jeżeli ktoś otrzyma sto, to oznacza to, że to jest zgodne z wynikiem doświadczalnym. To sto to jest coś, co możemy porównać, jednoznacznie nałożyć dwa zwinięte sznurki na siebie i powiedzieć, że one idealnie się pokrywają. Jeżeli się idealnie pokrywają, to otrzymaliśmy sto procent zgodności. Do tej pory te wielkości były znacznie niższe niż sto. Okazuje się, że w ostatnim przewidywaniu, czyli tzw. CASP14…
K.G.: Bo tak ten konkurs się nazywa?
J.S.: Tak, co dwa lata ma kolejny numerek. Puenta jest taka, że w tym roku dwie trzecie struktur AlphaFold 2 przewidział z dokładnością około dziewięćdziesięciu procent. I to było to wow, dostaliśmy jakość, która jest tak samo dobra jak doświadczalnie. Tu było jeszcze jedno wielkie wow – okazało się, że są takie znane przypadki, że na podstawie tych danych teoretycznych kilku grupom udało się poprawić dane doświadczalne. To znaczy, mając to przewidywanie teoretyczne, porównali to ze swoim eksperymentem i np. w jednym przypadku zauważyli, że jeden aminokwas jest po prostu źle wyznaczony, jego kierunek w przestrzeni jest inny, niż powinien być, więc teoria pozwoliła ulepszyć dane doświadczalne. A z kolei np. w przypadku tzw. białek membranowych – są to białka, które są zanurzone w membranie – jest znacznie mniej ich znanych struktur, ponieważ są one bardzo ciężkie do wyznaczania doświadczalnego. Zawsze stanowiły bardzo duże wyzwanie dla teoretyków. Okazało się, że ten model teoretyczny z AlphaFold 2 pozwolił znacznie lepiej rozwiązać strukturę. Więc w tej chwili aż trudno sobie wyobrazić, co będzie dalej.
K.G.: Będę cię o to za chwilę wypytywać, natomiast chciałabym, żebyśmy teraz trochę wróciły do podstaw. Wyobraź sobie, że my wszyscy tutaj, publiczność, nie do końca wiemy, jak białka mogą być istotne. Więc jeszcze raz, od początku. Białka to jest w zasadzie podstawa życia. One są w nas wszędzie, w każdej komórce. Co one takiego robią? Jakbyś np. podała takie najsłynniejsze białka, żebyśmy mogli sobie skojarzyć te nazwy, które na co dzień znamy, ale np. nie wiemy, że to są białka?
J.S.: Najsłynniejsze białka to też jest pojęcie względne. Teraz wszyscy się skupiamy np. na białkach z wirusa SARS-CoV-2. Myślę, że to są obecnie jedne z najsłynniejszych białek, dla których projektuje się inhibitory, czyli cząsteczki, które mogą blokować funkcje danego białka. Myślę, że najpierw trzeba powiedzieć, że białka to są takie podstawowe cząsteczki, elementarne cząsteczki, z których jesteśmy złożone. Składają się one z dwudziestu różnych aminokwasów, które są ułożone w łańcuch. Tak jak mamy kod PIN do banku, tak mamy cztery cyferki albo więcej. Każde z nas wie, że im więcej mamy tych cyferek, tym możemy je bardziej skomplikowanie ułożyć i to nasze zabezpieczenie do konta jest bezpieczniejsze. Można też tak pomyśleć o białkach, że to jest dwadzieścia literek, które ewolucja zaplanowała sobie, jak ułożyć, żeby otrzymać trójwymiarową strukturę. I ta trójwymiarowa unikalna struktura pełni daną funkcję biologiczną. Czyli np. mogą to być enzymy, które są odpowiedzialne za przeprowadzanie katalizy, czyli jakiejś reakcji chemicznej, obniżając ten próg przejścia wykonania reakcji chemicznej. Mogą być to też białka membranowe. Mamy różne białka, które są białkami sygnałowymi. Takim białkiem sygnałowym, które myślę, że cały czas jest bardzo znane, jest np. leptyna, która jest odpowiedzialna za otyłość. Ona wiąże receptor, czyli inne białko, które wysyła sygnał do mózgu, który nam mówi, czy mamy jeść, czy mamy przestać jeść.
K.G.: Różne są te słynne białka, ale ja wiem, jakie jest najsłynniejsze. Moim zdaniem.
J.S.: Jakie?
K.G.: Kolagen. Żebyśmy były piękne i wiecznie na skórze twarzy napięte. [śmiech]
J.S.: No tak, ale myślę, że jakbyś zapytała sportowca, jakie jest dla niego najsłynniejsze białko, to by powiedział, że tytyna, która jest odpowiedzialna za mięśnie i za ich pracę.
K.G.: To ja teraz powiem: hemoglobina. O tym na pewno każdy słyszał.
J.S.: Tak, to prawda. Wydaje mi się, że dla każdego w danym momencie życia inne białko jest ważne. Hemoglobina oczywiście jest podstawowym białkiem, ale myślę, że także białka, które są związane z otyłością, z chorobą Parkinsona – mówi się w skrócie „białko UHCL”, które nie do końca wiemy, jak działa, ale wiemy, że mutacja, czyli wymiana pojedynczych aminokwasów, może prowadzić do choroby Parkinsona albo do innych chorób neurogenetycznych. Pozwolę sobie tutaj nawiązać do tego ostatniego CASP-u. Jeżeli mielibyśmy projektować jakieś leki albo zrozumieć pojedynczą funkcję danego białka i jakie ono ma znaczenie dla ludzi, w jaki sposób może nam to pomóc w zwalczaniu chorób, projektowaniu leków, to AlphaFold daje nam w pewien sposób odpowiedź, ponieważ on przewiduje strukturę z rozdzielczością eksperymentalną. Czyli teraz na podstawie tej jakości przewidzianej struktury możemy rozpocząć różne badania teoretyczne, jak projektowanie leków. Wiemy, że w tej chwili mnóstwo firm na świecie ściga się w projektowaniu szczepionki, która nie będzie miała tylko dziewięćdziesięciu procent skuteczności, ale sto procent. Więc tu puentą jest, że AlphaFold przewidział strukturę tak dokładnie, że nawet małe niuanse, które są np. w małych pętelkach w białku, są poprawnie wymodelowane. Jest około stu białek, dla których w danych zawodach członkowie grup starają się przewidzieć strukturę. On otrzymał tę jakość w dziewięćdziesięciu procentach dla dwóch trzecich. Ale i tak to otwiera kilkaset drzwi do różnych badań teoretycznych, które są znacznie tańsze niż badania eksperymentalne. Jeśli spytałabyś mnie, w jaki sposób udało im się przewidzieć taką jakość, to powiedziałabym ci, że nie wiem, i tak naprawdę nikt dokładnie nie wie. Jest podany schemat, w jaki sposób oni to otrzymali.
K.G.: Pytanie, czy oni sami wiedzą, czy wie to tylko ta sztuczna inteligencja.
J.S.: Oni opracowali technikę, tylko nie powiedzieli, jakie są jej szczegóły. Tak samo jest też w różnych innych metodach. Dopóki nie ukaże się praca naukowa, która opisuje szczegóły, nie możemy tego powielić. Ale w tym przypadku ich praca jest niepodważalna. Ich wynik jest porównywalny z doświadczeniem. To znaczy, mamy tę czarną kulę, oni znają jej algorytm, ale my dostaliśmy wynik, który porównujemy jednoznacznie z wynikiem doświadczalnym. Więc tutaj nie należy upatrywać żadnych magicznych rzeczy. Tak samo nie ma żadnych magicznych rzeczy w przewidywaniu leków, ale ten algorytm nie jest jeszcze do końca znany. Algorytm z zeszłego roku z CASP13, który był używany, był nazwany Alpha. Ten algorytm jest publiczny. Jest zdeponowany na tzw. GitHubie. Każdy, kto się zajmuje informatyką, deponuje tam dane, więc każdy może sobie ściągnąć ten kod i spróbować odtworzyć dane. Prawdopodobnie będzie tak także w tym przypadku, ale oni chcą to najpierw zapewne opisać w jednym z najlepszych pism, czyli np. w „Nature” jako wytłumaczenie i podanie składowych elementów, w jaki sposób udało im się do tego dojść. Jeszcze raz powtarzam, że puentą jest to, że do projektowania leków, jeżeli mówimy o znaczeniu białek, czyli chcemy wiedzieć, jak działa hemoglobina, kolagen, potrzebne nam są dane, które są tzw. eksperymentalnej rozdzielczości. Rozdzielczość eksperymentalna to jest około dwóch angstremów, jeżeli mamy być bardzo dokładni. To jest miara, której się używa do porównywania np. dwóch struktur. Jeżeli nałożymy na siebie dwie struktury, które są linią prostą, to łatwo je na siebie nałożyć i powiedzieć, czy się idealnie pokrywają. Jeżeli są bardziej zawiłe, to może trzeba użyć bardziej zawiłego wielomianu, żeby dopasować jedną krzywą do drugiej. Summa summarum to dopasowanie jest mniejsze niż dwa angstremy nawet dla białek, które mają około stu aminokwasów. Więc to jest naprawdę wow.
K.G.: To znaczenie struktury przestrzennej jest fascynujące. No bo mamy sobie białko, które składa się z iluś tam aminokwasów, tych literek czy koralików, żeby było to łatwiejsze do wyobrażenia. Koralików o różnych kolorach. No i chemicznie to jest to jedno i to samo, tak to przynajmniej rozumiem. Więc jak to jest możliwe, że to, w jaki sposób to białko się składa, w jaki sposób ten sznurek się układa, ma znaczenie dla jego funkcjonowania biologicznego? Nie bardzo wiem, dlaczego właśnie ta struktura jest taka istotna, skoro gdyby to rozpisać na kartce: tutaj jest taki związek, tu jest taki związek, tu jest taki pierwiastek, no to to wszystko jest to samo. A tu się okazuje, że nagle struktura jest ważna biologicznie. Ty się tym bardzo mocno zajmowałaś, udowodniłaś też, że białka mogą się tak splątywać, zaplątywać. Z czego to wynika? O co tutaj chodzi? Wiecie czy po prostu przyjmujecie, że tak jest i staracie się jakoś wykorzystać tę wiedzę, ale dlaczego ta struktura jest istotna, to tego nie wiemy?
J.S.: Dlaczego struktura jest istotna? Myślę, że możemy powiedzieć, upraszczając, dając przykład klucza, który pasuje do zamka. Jeżeli chcemy gdzieś wejść i te drzwi są zamknięte, to musimy mieć idealnie dopasowany klucz, który nam ten zamek otworzy. Tak samo jest z białkami, np. białka enzymatyczne muszą dopasować do siebie inną malutką cząsteczkę, żeby np. otworzyć jej funkcje, żeby mogła się ona idealnie dopasować, jak puzzle. Te puzzle są, że tak powiem, łatwe w układaniu, można powiedzieć, że ich brzegi są zazwyczaj proste, tylko mają małe kieszenie i do tych kieszeni dopasowujmy sobie drugi kawałek. Te kształty nie zawsze są idealne, proste. W przypadku białek są to poszczególne atomy, które muszą się do siebie idealnie dopasować. Jeżeli mamy puzzle, to te krawędzie są gładkie i tu wszystko bardzo ładnie pasuje. Tak jest w naszym świecie, gdzie możemy mówić o centymetrach, ale teraz możemy zejść kilka wymiarów niżej do angstremów. Tutaj mamy powierzchnie, które nie są gładkie. Jedna cząsteczka wody, która będzie inaczej ułożona, może zdecydowanie wpłynąć na to, że to białko będzie w stanie pełnić swoją funkcję biologiczną, czyli zadokować albo umieścić ten klucz w kieszeni. I dopiero, jeżeli umieścimy ten klucz w kieszeni, to to białko zacznie funkcjonować, zacznie wysyłać sygnał do mózgu, przeprowadzać reakcję enzymatyczną, w przypadku białek membranowych transportować jony, które są kluczowe dla funkcjonowania komórki. Tu jest ten problem o wiele bardziej złożony. Wiemy, że struktura jest kluczowa dla funkcji biologicznej, ale pytanie, w jaki sposób otrzymujemy tę strukturę – pozwolę sobie zażartować, że AlphaFold wie lepiej niż my obecnie. Jest to jeden z dogmatów biologii czy biofizyki strukturalnej, paradoks długo już postulowany, w jaki sposób ta sekwencja aminokwasów wie, do jakiej struktury się zwinąć. Tu sobie żartuję, że sztuczna inteligencja rozwiązała ten problem szybciej niż my. To znaczy, my jesteśmy w stanie przewidywać struktury białek, ale jak pokazują ostatnie zawody, sztuczna inteligencja robi to lepiej niż jakiekolwiek inne grupy. A tu warto moim zdaniem wspomnieć o takich osobach jak Krzysztof Ginalski albo Andrzej Koliński, którzy byli jednymi z pionierów w przewidywaniu struktur przestrzennych białek, i osiągali niesamowite sukcesy kilka lat temu w jednych z pierwszych zawodów. Są to znakomici naukowcy, którzy myślę, że mieli w sobie coś ze sztucznej inteligencji. Oni to robili bardziej ręcznie, to znaczy, używali zaawansowanych metod informatycznych, ale tam był ten pierwiastek ludzki. Oni potrafili spojrzeć na struktury, których mieli na koniec z pięćdziesiąt, i dokonać tego, co się robi w sztucznej inteligencji. Neuronowe połączenia w ich mózgach potrafiły powiedzieć: ta tak, a ta nie. Więc myślę, że tu jest ten pierwiastek ludzki. Myślę, że nie zawsze wiedzieli, dlaczego ta, ale coś im podpowiadało, że to jest ta. To znaczy, potrafili tak to wypatrzeć, że przewidywali dobrze.
K.G.: Czyli im lepszą mamy wiedzę na temat tej struktury białek, tego, w jaki sposób te sznureczki się składają, tym lepiej rozumiemy biologię, w tym biologię człowieka, tym lepiej rozumiemy np. różne przyczyny chorób, a więc możemy lepiej je leczyć. To jest rzecz, która oczywiście nas wszystkich zwykle interesuje najbardziej. Czyli wracamy do tych leków, o których mówiłaś. Dzięki lepszemu rozumieniu tej struktury będziemy mogli projektować nowe leki? Właśnie tak na kartce, a potem je od razu wysyłać do firm farmaceutycznych, żeby je produkowały? Tak to może wyglądać?
J.S.: Tak to jeszcze nie będzie, bo zawsze jednak trzeba się upewnić, czy działa. Tu do projektowania leków podstawową rzeczą jest najpierw znajomość struktury. To znaczy, jest to zupełnie nieopłacalne – wrzucać kilkanaście związków do roztworu i patrzeć, który zadziała. Tak się tego nie robi. Do projektowania leków jest potrzebna znajomość struktury przestrzennej. Jeżeli znamy tę strukturę, to możemy robić tzw. screening albo sprawdzać siedemdziesiąt milionów związków, które są w tej chwili dostępne w różnych bazach leków zatwierdzonych np. przez Amerykańską Agencję ds. Żywności i Leków albo na podstawie innych znanych baz związków możemy je automatycznie dokować. I tutaj także się używa algorytmów do sztucznej inteligencji, które próbują zadokować małą cząsteczkę do dużej. Jeżeli nam się uda zadokować tę cząsteczkę, to możemy powiedzieć, czy np. wyhamowaliśmy funkcję danego białka, czy ona nadal jest aktywna. Często się zdarza też tak, że jedna cząsteczka niestety może pasować także do innego białka. Innymi słowy, to oznacza, że może być toksyczna. Chcielibyśmy znaleźć taką cząsteczkę, która by zablokowała specyficznie tylko jedno białko. Nie działa nam na wszystkie nasze inne białka i stąd mogą być różne efekty uboczne, ale działa selektywnie, działa tylko na jedno białko i blokuje tylko jedno białko, które jest np. dla nas szkodliwe albo jest w tej chwili uszkodzone. Chcielibyśmy, żeby przestało się namnażać, albo nastąpiła w nim jakaś mutacja i chcielibyśmy zrozumieć, w jaki sposób ona działa.
K.G.: Czy to jest tak, że AlphaFold rozwiązało główną zagadkę i w zasadzie już konkurencja się zakończyła, wiemy wszystko o strukturach białek, czy też dostaliśmy takie narzędzie i po prostu będziemy teraz je wykorzystywać? Czy może jednak są jeszcze takie duże zagadki dotyczące białek, które czekają na wyjaśnienie, lub na narzędzia, które ułatwią jak najlepsze rozumienie tych tajemnic?
J.S.: Myślę, że to był krok milowy. Na pewno dobrze by było dojść do tego, żeby to było nie dwie trzecie białek, tylko wszystkie. Pozwolę sobie wtrącić inny wątek – niedawno udało się wejść na K2 i myślę, że to był niesamowity wyczyn. Każdy, kto był w górach powyżej pięciu czy sześciu tysięcy, wie, że jest to niemałe wyzwanie i trudno się wtedy poruszać. Dla mnie niesamowite było to, że oni na siebie poczekali te jedenaście metrów przed szczytem, bo też nieraz byłam w takiej sytuacji i wiem, że to nie jest łatwe, żeby sobie odmówić i poczekać na ekipę. Więc chylę czoła przed wejściem na K2 i przed AlphaFold. To jest niesamowite osiągnięcie. Tak samo jak niesamowitym osiągnięciem jest wejście na K2. Czy ono było z tlenem, czy bez tlenu, uważam, że to jest niepodważalne osiągnięcie. I tak samo uważam, że niepodważalnym osiągnięciem są wyniki AlphaFold 2. Mimo tego, że to nie jest sto procent, tylko te dwie trzecie. Zostaje jedna trzecia białek, które się wymknęły.
I pytanie, dlaczego się wymknęły, czy jest w nich coś specyficznego, co mogło spowodować to, że się wymknęły. Tu mogę powiedzieć, że nasze badania pokazują, że to są częściowo białka z węzłami. Okazuje się, że te, które mają tzw. nietrywialną topologię, czyli takie, które mają na sznurku zawiązany węzełek, jakoś nie do końca się dobrze udało przewidzieć. Więc trochę się cieszę, że pozostało miejsce konkretnie dla naszych badań. Wydaje mi się, że jest kilka rzeczy, które nadal zostają do rozwiązania i które są kluczowe do tego, żeby np. projektować leki. To jest oddziaływanie białko-białko. A więc nie tylko przewidzenie struktury przestrzennej pojedynczego białka, czyli jednego sznurka, ale przewidzenie, jak jeden sznurek oddziałuje z drugim. I dopiero, jak to będziemy mieli, to znaczy, jak będziemy wiedzieli, jak dwa sznurki ze sobą oddziałują, czyli duża i mała cząsteczka albo dwie duże, to to by była taka kropka nad i, to otworzyłoby sto procent możliwości projektowania leków.
Drugim tematem, który tutaj w ogóle nie był podejmowany w przewidywaniu struktury białek, jest to, w jaki sposób białko się zwija. To jest to, o co pytałaś wcześniej, to znaczy, w jaki sposób z tej sekwencji aminokwasów dochodzimy do tej trójwymiarowej struktury. I tu warto wspomnieć, że pewnym przełomem były także badania prowadzone przez superkomputer Anton. D. E. Shaw jakiś czas temu pokazał, że jest w stanie zwinąć mały peptyd, i teraz te badania postępują dalej. To znaczy, że jest w stanie opracować tzw. kinetykę, czyli kolejne ruchy, które są potrzebne, żeby taki prosty odcinek pozginać w odpowiedni sposób, żeby na koniec otrzymać unikalną strukturę trzeciorzędową.
K.G.: Taką, która działa biologicznie – to jest ważne. Bo poskładać ją można w różny sposób, ale kluczowe jest to, żeby ją poskładać tak, jak tego chce natura, tak?
J.S.: Dokładnie. Tu można by było sobie wyobrazić, że są tysiące, miliony takich ścieżek, za pomocą których można by było ją składać, ale okazuje się, że nie, że ewolucja tak to zaprojektowała, że jest kilka, a zazwyczaj może nawet jedna główna ścieżka, która nam mówi, jak np. dojść z jednego miejsca Warszawy do drugiego. Możemy iść na wprost, w lewo, w prawo i tych ścieżek może być naprawdę tysiące, i jeszcze małych uliczek. Jest jeszcze pytanie, czy metrem, czy autobusem. Ale ewolucja tak to zaprojektowała, że to jest zazwyczaj jedna ścieżka, może z małymi zakrętami.
Pytanie, czy AlphaFold będzie w stanie kiedyś rozwiązać problem zwijania białek. Tu są inne grupy, np. warto wymienić grupę Cecilii Clementi i Franka Noé z instytutu w Berlinie, którzy ostatnio opublikowali bardzo dobre prace w „Nature” na temat zwijania pojedynczego peptydu czy pojedynczego białka. Chyba trzeba to niestety nazwać peptydem, bo ma, o ile dobrze pamiętam, około dziesięciu, piętnastu aminokwasów. Zazwyczaj takie małe sznurki nazywamy peptydami, nie białkami. Oni skorzystali z tych danych, które były przeliczone na superkomputerze. Następnie na podstawie tych danych wytrenowali model sztucznej inteligencji, żeby odtworzyć ścieżkę zwijania. To jest kolejny etap, który moim zdaniem jest przełomowy. Ta praca ukazała się chyba już ponad rok temu i do tej pory nie pojawiły się prace, które byłyby w stanie zwinąć coś większego. Więc to też pewna zagadka, czy to jest kwestia nie tyle tego, na ile algorytm jest zawiły, ile ogromu danych, które są potrzebne do przerobienia i wytrenowania układu, żeby można było zwinąć białko. Wydaje mi się, że to są takie dwa elementy. Trzecim elementem może być np. konkretne przekraczanie bariery energetycznej. Tu warto powiedzieć, że np. te algorytmy sztucznej inteligencji bardzo dobrze sobie radzą w przypadku reakcji chemicznych, gdzie jest to mały układ kilku atomów i sztuczna inteligencja jest tutaj ostatnio powszechnie wykorzystywana do uczenia i wykonywania takich małych reakcji chemicznych.
K.G.: Wygląda na to, że dla twojej dziedziny szykują się wspaniałe dziesięciolecia, jeśli chodzi o możliwość rozwoju.
J.S.: Myślę, że tak. To jest coś, co nas fascynuje i myślę, że trzeba korzystać z dobrodziejstw rozwoju nauki. To pokazuje także, jak ważna jest interdyscyplinarność, bo myślę, że każdy sobie zdaje sprawę z tego, że AlphaFold jest powołany przez firmę Google, jeżeli się nie mylę chyba w 2014 roku, ale nie jestem pewna. Więc nie byli to stricte biolodzy. Byli to informatycy, matematycy. W tym zespole są oczywiście biolodzy, bo trzeba takie struktury rozumieć, ale to pokazuje, że pytanie, czy jesteś fizykiem, chemikiem, czy biologiem, trochę się zatraca.
K.G.: Ty przecież jesteś najlepszym przykładem fizyka. Zdaje się, że to był twój pierwszy kierunek wykształcenia.
J.S.: Tak, później była chemia, a teraz dużo się zajmuję też biologią, bo fascynuje mnie pogranicze zastosowania metod fizycznych, chemicznych i informatycznych do zrozumienia procesów biologicznych. Ale wydaje mi się, że właśnie takie interdyscyplinarne podejście i umiejętność korzystania z różnych dziedzin jest tym, co nas prowadzi do takich przełomowych odkryć, jakie otrzymał AlphaFold. To jest zespół, który musiał umieć współpracować, musiał mówić językiem prostym, komunikować się i zadawać sobie pytania, które normalnie by się wydawało, że są abstrakcją.
K.G.: Drodzy słuchacze, naprawdę pilnujcie strefy biologii, bo XXI wiek jest i będzie wiekiem biologii, na pewno się jeszcze dowiemy mnóstwa rzeczy. Również dzięki takim znakomitym badaczkom, jaką jest doktor habilitowana Joanna Sułkowska, profesor Uniwersytetu Warszawskiego i kierownik Interdyscyplinarnego Laboratorium Modelowania Układów Biologicznych. Bardzo ci dziękuję, że znalazłaś czas dla Radia Naukowego.
J.S.: Bardzo dziękuję. Myślę, że to będzie niesamowity rok – zarówno ten, jak i kolejne dla badań naukowych. Bo tylko jeszcze wspomnę na koniec, że sztuczna inteligencja już zrobiła przecież niesamowity postęp w nauce języka. Algorytmy do rozpoznawania mowy to jest coś, od czego się zaczęło. Więc myślę, że w każdej dziedzinie będzie to przełom.
K.G.: Pełna zgoda. Jeszcze raz dziękuję, Joanno. Wszystkiego dobrego, powodzenia w badaniach.
J.S.: Dziękuję bardzo. Do usłyszenia.
Przygotowanie każdego odcinka to wiele godzin pracy, dlatego jeśli spodobał wam się ten podcast, możecie zajrzeć na serwis Patronite – patronite.pl/radionaukowe i tam wspomóc moją pracę, a także rozwój Radia Naukowego. Dzięki.

Fizyczka, chemiczka, kierowniczka Interdyscyplinarnego Laboratorium Modelowania Układów Biologicznych w Centrum Nowych Technologii Uniwersytetu Warszawskiego. Zainteresowania badawcze: modele teoretyczne krajobrazu energetycznego białek, metody analityczne i narzędzia bioinformatyczne w przewidywaniu struktur białkowych, właściwości mechaniczne białek, degradacja i translacja.